Visualisation des Régions Conservées
dans les structures en 3D des Protéines
|
|
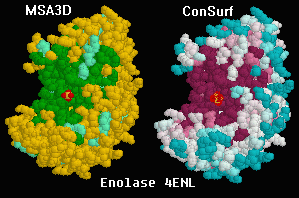
Les régions sombres indiquenr la conservation du site
catalytique de l'énolase.
|
Les régions conservées (au taux de mutations inférieur à la moyenne)
Peuvent visualisées sur la structure 3D d'une protéine
en aplliquant des coloris basés sur l'alignement de séquences multiples.
Ces régions signalent en général des zones dont l'importance
fonctionnelle est cruciale.
En Décembre 2001, Glaser, Ben-Tal, Pupko et
Martz ont mis en place le
Serveur ConSurf. ConSurf génère automatiquement un
alignement de séquences multiples de protéines (ou en accepte un préalablement réalisé),
puis génère automatiquement un arbre phylogénétique,
et applique des couleurs réprésentant l'indice de conservation
de chaque résidu de la structure 3D de la protéine. Les résultats sont
affichés dans Protéine Explorer.
Précédemment, début 2000, Protéine Explorer
proposait l'ASM3D, qui acceptait des alignements de séquences multiples de protéines
fournis par l'utilisateur, et les utilisaient pour colorer la structure 3D de la protéine. L'ASM3D
ne distingue que 3 catégoris de résidus: identique, similaire, and différent.
ConSurf est plus facil à utiliser que l'ASM3D, parce qu'il est complétement automatisé
et qu'il fournit tout ce qui est nécessaire sur un seul site.
Plus important, les algorithmes employés par ConSurf sont beaucoup plus sophistiqués.
Dans presque tus les domaines, ConSurf est supérieur à L'ASM3D. C'est pourquoi nous vous recommandons
d'utiliser
ConSurf pour visualiser les régions conservées dans les structures en 3D des protéines.
La documentation ci dessous, décrit l'ASM3D, conserve un intérêt principalement
historique.
Alignement de Séquences Multiples
en 3D dans Protéine Explorer (ASM3D): L'Enolase
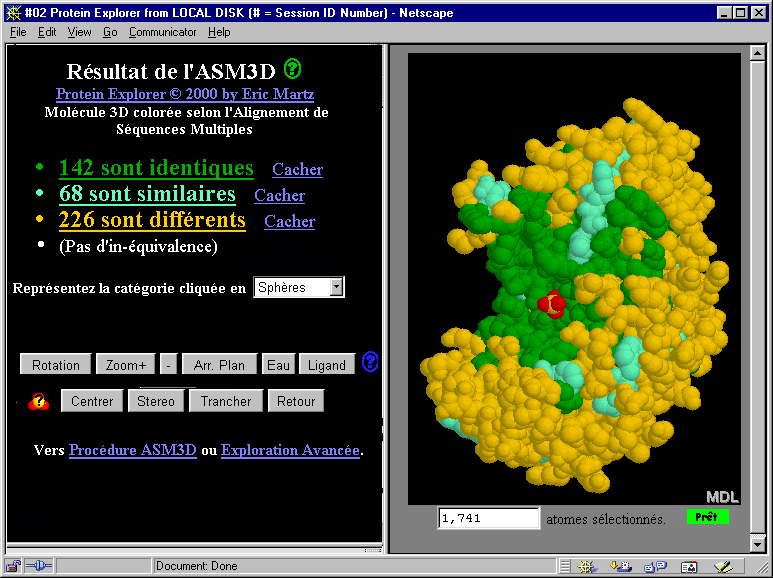
Un alignement de multiples séquences d'une protéine a été
réalisé pour l'énolase. Les commandes de l'ASM3D de
Protéine Explorer ont attribué des couleurs correspondant à
l'identité, à la similarité, ou à la différence
des acides aminés alignés, comme l'indique le listing
de l'alignement de Protéine Explorer. L'alignement comprend des
protéines provenant Eubacteries, d'Archebacteries, et d'Eucaryotes (Drosophile,
Levure, et Homme). Malgré l'extraordinaire diversité de l'échantillonage
et de la durée de l'évolution, tous les résidus du
site catalytique de l'enzyme sont identique. (Le site catalytique est marqué
par la présence d'un ion sulfate (en rouge) qui s'y est lié dans
cette molécule, 4enl.)
Si vous avez installé Chime, vous pouvez voir cette molécule en rotation dans une fenêtre
interactive. Sinon,
téléchargez Chime et installez le cela ne prend que quelques
minutess.
Les liens dans le cliché ne fonctionnent pas puisqu'il s'agit
uniquement d'un cliché.
Remerciements à Paul Stothard qui a crée une grande
partie du code de l'ASM3D, et à Garry Duncan qui a fourni cet alignement.
 Comment trouver l'ASM3D après avoir lancer Protéine Explorer? A
partir de la PremièreVue, cliquez "Explorer Plus", puis vous verrez un
lien vers "Exploration avancée", ou vous trouverez l'ASM3D dans le menu
proposé. L'alignement de l'enolase est intégré en démonstration
à Protéine Explorer avec 2 autres exemples.
Comment trouver l'ASM3D après avoir lancer Protéine Explorer? A
partir de la PremièreVue, cliquez "Explorer Plus", puis vous verrez un
lien vers "Exploration avancée", ou vous trouverez l'ASM3D dans le menu
proposé. L'alignement de l'enolase est intégré en démonstration
à Protéine Explorer avec 2 autres exemples.
Ce lien spécial ASM3D-Enolase
lance directement Protéine Explorer(si votre installation est correcte),
Charge l'enolase par Internet (il faut être connecté), et passe
automatiquement de la PremièreVue à l'ExplorationAvancée,
qui donne accès à l'ASM3D.
Un tutoriel détaillé accompagne
l'ASM3D et explique comment construire un alignement et comment l'utiliser pour
colorer la molécule étudiée.


